Les fonctions de survie sous R
Introduction
Quelques distributions
Estimation de Kaplan-Meier
Modèles à temps discret
Modèle de régression paramétrique
Proportional Hazard (PH) models
Accelerated Failure Time (AFT) models
Autres modèles
Cox Proportional Hazard
Introduction
On s'intéresse ici à des variables de survie : une variable de survie, c'est à peu près comme une variable quantitative, mais elle peut prendre des valeurs numériques précises, comme "13 ans", mais aussi des valeurs moins précises, du genre "plus de 15 ans".
On qualifie de "valeurs censurées" ces valeurs moins précises. On les note généralement ainsi :
10+ Plus de 10 (donnée censurée à droite) 5- Moins de 5 (censurée à gauche) [5,10] Entre 5 et 10
On les rencontre par exemple quand on regarde la durée de survie après un cancer : si l'étude dure 10 ans, on pourra donner la durée de survie de tous les patients qui sont morts, mais pour les autres, on pourra juste dire que leur durée de survie est supérieure à 10 ans. La censure à gauche correspond à des évènements qui ont eu lieu avant le début de l'étude (mais tous les individus ne rentrent pas dans l'étude en même temps). A FAIRE : donner un exemple concret de censure à gauche. La censure par intervalle se rencontre par exemple quand on constate l'apparition d'un symptome lors d'une visite médicale : il est apparu depuis la visite précédente, mais on ne sait pas exactement quand.
Voici d'autres exemples : durée de survie d'un patient, durée de survie d'un mariage, durée de survie d'une entreprise, durée de fonctionnement sans panne d'un produit manufacturé, durée pendant laquelle un produit reste suffisemment populaire pour continuer à être produit, durée de survie d'un enseignant en lycée (jusqu'à sa démission, son internement en hopital psychiatrique, son suicide), durée jusqu'à l'arret de prise d'un médicament, etc.
On peut aussi voir les variables de survie comme des variables qualitatives binaires (celles qu'on cherche à prédire dans la régression logistique) indiquant la survenue ou non d'un évènement, auxquelles on a ajouté l'instant de survenue de cet évènement. Les valeurs censurées correspondent à << l'évènement ne s'est pas produit >> : dans les exemples précédents, cela revient à remplacer les valeurs << Vivant / Mort >> par << Vivant / Date de la mort >>.
Les variables de survie peuvent avoir plusieurs valeurs : par exemple sain, malade et mort (si un patient guérit entre deux maladies, ça devient encore plus compliqué). Dans ces situations plus complexes, on parle parfois de données transitionnelles (car on s'intéresse à la transition d'un état vers un autre) ou de "spell duration data".
On peut aussi voir une donnée de survie comme une série temporelle qualitative, dans laquelle on s'intéresse aux transitions d'un état à l'autre.
A FAIRE :
Competing risk models Un état de départ et plusieurs états absorbants (exemple : passage du chomage à un emploi ou du chomage à une sortie du marché de l'emploi ; autre exemple : concurrence entre mort du cancer et mort de maladie cardiovasculaire).
Si T est une variable de survie, sa fonction de survie est
S(t) = P(T>t) = 1-F(t).
Par exemple, si T désigne le nombre d'années de survie après la diagnostic d'un cancer, S(t) est la probabilité de survivre au moins t années.
Sa "hazard function", ou "hazard rate" (je ne sais pas comment on dit en français : fonction de risque ?) est
P( t < T <= t + u | T > t )
lambda(t) = lim -----------------------------
u -> 0 u
dF/dt
= -------
S
d ln S
= - --------
dt
La formule ressemble beaucoup à la définition de la densité, mais on a ajouté la condition "T>t".
A la différenciation près, c'est très semblable à la différence entre l'espérance de vie à la naissance et l'espérance de vie à 60 ans, ou la différence entre la probabilité de mourir entre 12 et 13 ans et la probabilité de mourir entre 12 et 13 ans sachant que le patient a déjà survécu 12 ans.
On rencontre aussi parfois la "cumulative hazard function", primitive de la précédente, qui représente le risque cumulé.
Lambda(t) = - ln S(t)
Quelques distributions
Elle correspond à une "hazard function" constante,
lambda(t) = lambda Lambda(t) = lambda t S(t) = exp( - lambda t ) op <- par(mfrow=c(3,1)) n <- 20 lambda <- rep(2,n) x <- seq(0,2,length=n) plot(lambda ~ x, type='l', main=expression(lambda)) plot(lambda*x ~ x, type='l', main=expression(Lambda)) plot(exp(-lambda*x) ~ x, type='l', main="S") par(op)

Elle correspond à une "hazard function" de la forme
Lambda(t) = a * t ^ gamma S(t) = exp( - a * t ^ gamma ) op <- par(mfrow=c(3,1)) n <- 20 alpha <- 1 g <- rep(2,n) x <- seq(0,2,length=n) plot(g * alpha * x^(g-1) ~ x, type='l', main=expression(lambda (gamma==2))) plot(alpha * x^g ~ x, type='l', main=expression(Lambda)) plot(exp(-alpha*x^g) ~ x, type='l', main="S") par(op)
A FAIRE : expliquer
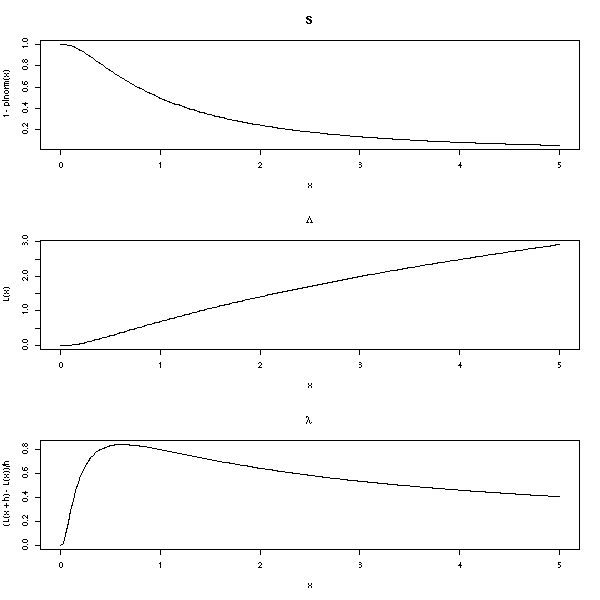
op <- par(mfrow=c(3,1))
n <- 200
x <- seq(0,5,length=n)
plot(1-plnorm(x) ~ x, type='l', main="S")
L <- function (x) { -log(1-plnorm(x)) }
plot(L(x) ~ x, type='l', main=expression(Lambda))
h <- .001
plot( (L(x+h)-L(x))/h ~ x, type='l', main=expression(lambda))
par(op)
Weibul: lambda = alpha * t^(alpha-1) * exp(+X béta)
???
A FAIRE : il y a une variable prédictive, qui n'a pas encore sa
place ici...
log-logistic model:
1
S(t) = -------------------------- with lambda = exp(- X béta)
1 + (lambda t)^(1/gamma)
Gompertz: lambda = exp( exp(+X béta) + gamma t )
(important)
Generalized Gamma
(généralise les modèles précédents: kappa=1:Weibul,
kappa=0:lognormal, kappa=sigma:gamma)
Il y a aussi des modèles à temps discret :
logistic complementary log-log
Estimation de Kaplan-Meier
S'il n'y a pas de censure, on peut estimer la fonction de survie ainsi :
Card( i : T_i>t )
S(t) = -------------------
n
En présence de censure, on tient compte des données censurées jusqu'à ce qu'on arrive à leur valeur, et après on les oublie. Plus précisément, si le temps est discret :
S(t0) = P(vivant à t=t0 | vivant à t=t0-1) * P(vivant à t=t0-1)
Par exemple, la fonction de survie de la série
1 2 2 2+ 3+ 3+ 3+ 4 4 4 4 ... (100 individus)
se calcule ainsi :
temps sujets morts censures p1 p2 S=p1*p2 ------------------------------------------------------ 1 100 1 0 99/100 1 .99 2 99 2 1 97/100 .99 .9603 3 96 0 3 96/100 .9603 .9219 4 93 3 0 90/100 .9219 .8297
Les valeurs de S sont des produits de plus en plus gros :
S(0) = 1 S(1) = 1 * 99/100 S(2) = 1 * 99/100 * 97/100 S(3) = 1 * 99/100 * 97/100 * 96/100 S(4) = 1 * 99/100 * 97/100 * 96/100 * 90/100 etc.
On peut donner une formule générale :
di
S(t) = Produit ( 1 - ---- )
i tel que ti<t ni
On peut calculer des intervalles de confiance en supposant que Lambda (qui vaut -log(S)) est normale.
A FAIRE : comment a-t-on la variance de Lambda ???
Il existe d'autres estimateurs de la fonction de survie, par exemple celui d'Altschuler--Nelson--Aalen--Flemming--Harrington :
di
Lambda(t) = Somme ------.
i tel que ti<t ni
Ces estimateurs ont une variance élevée : si on a un modèle raisonnable, il vaut mieux préférer un estimateur paramétrique obtenu par le maximum de vraissemblance (on peut signaler que l'estimateur de Kaplan--Meier correspond à un maximum de vraissemblance (non paramétrique)).
La plupart des fonctions sont dans le paquetage "survival".
La commande Surv permet de créer des variables de survie.
> Surv(c(1,2,2,2,3,3,3,4,4,4,4), + c(1,1,1,0,0,0,0,1,1,1,1)) [1] 1 2 2 2+ 3+ 3+ 3+ 4 4 4 4
La commande survfit permet de calculer la fonction de survie.
> x <- Surv(c(1,2,2,2,3,3,3,4,4,4,4),
+ c(1,1,1,0,0,0,0,1,1,1,1))
> survfit(x)
Call: survfit(formula = x)
n events rmean se(rmean) median 0.95LCL 0.95UCL
11.000 7.000 3.364 0.322 4.000 Inf Inf
> summary(survfit(x))
Call: survfit(formula = x)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
1 11 1 0.909 0.0867 0.754 1
2 10 2 0.727 0.1343 0.506 1
4 4 4 0.000 NA NA NA
ou de la tracer
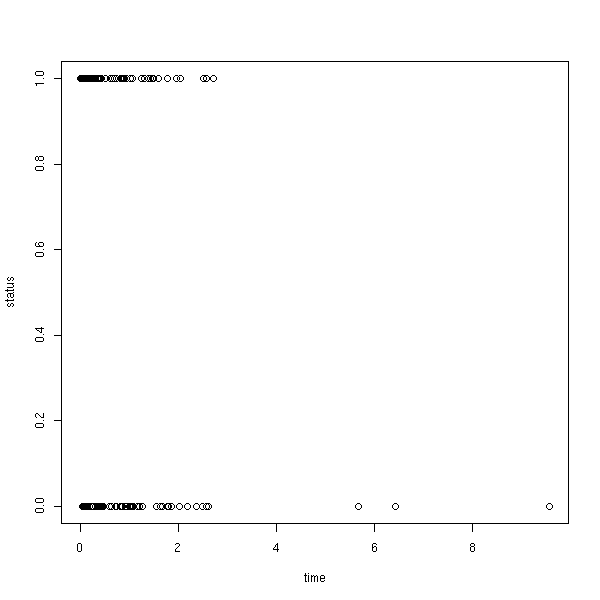
set.seed(87638) library(survival) n <- 200 x <- rweibull(n,.5) y <- rexp(n,1/mean(x)) s <- Surv(ifelse(x<y,x,y), x<=y) plot(s) # peu informatif
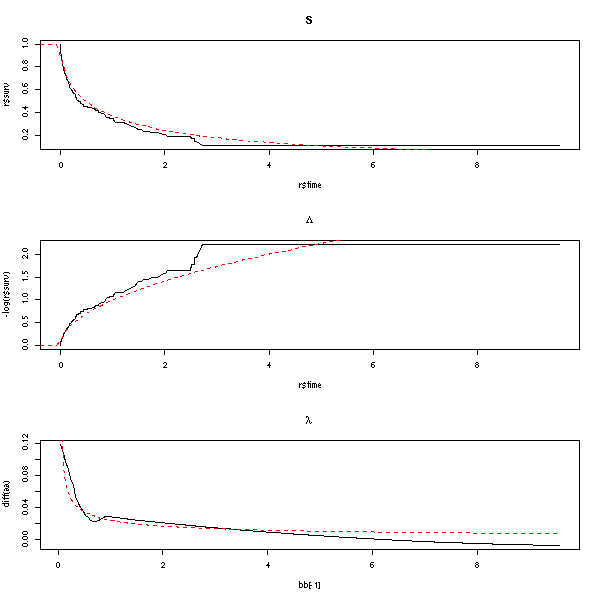
plot(survfit(s))
lines(survfit(s, type='fleming-harrington'), col='red')
r <- survfit(s)
lines( 1-pweibull( r$time, .5 ) ~ r$time, lty=3, lwd=3, col='blue' )
legend( par("usr")[2], par("usr")[4], yjust=1, xjust=1,
c("Kaplan-Meier", "Fleming-Harrington", "Fonction de survie théorique"),
lwd=c(1,1,3), lty=c(1,1,3),
col=c(par("fg"), 'red', 'blue'))
title(main="Fonction de survie (estimation de Kaplan-Meier)")

op <- par(mfrow=c(3,1)) r <- survfit(s) plot(r$surv ~ r$time, type='l', main="S") curve( 1-pweibull(x,.5,1), col='red', lty=2, add=T ) plot(-log(r$surv) ~ r$time, type='l', main=expression(Lambda)) curve( -log(1-pweibull(x,.5,1)), col='red', lty=2, add=T ) # Avant de dériver, on commence par lisser Lambda a <- -log(r$surv) b <- r$time library(modreg) l <- loess(a~b) bb <- seq(min(b),max(b),length=200) aa <- predict(l, data.frame(b=bb)) plot( diff(aa) ~ bb[-1], type='l', main=expression(lambda) ) aa <- -log(1-pweibull(bb,.5,1)) lines( diff(aa) ~ bb[-1], col='red', lty=2 ) par(op)
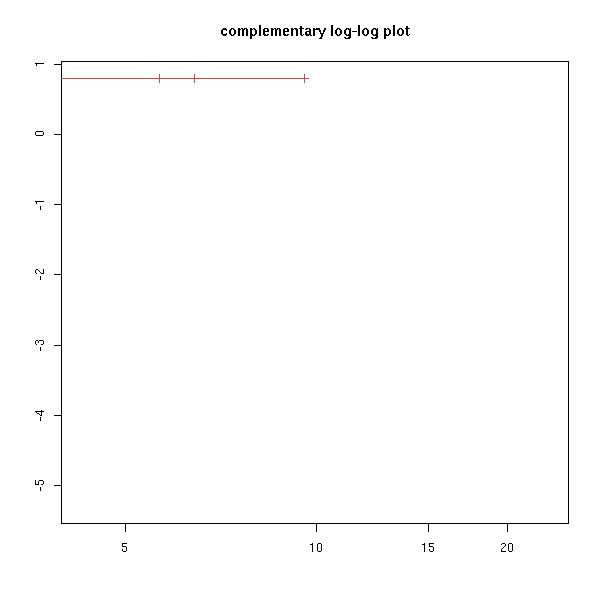
Remarque : on peut obtenir certains des graphiques ci-dessus en modifiant l'option "fun".
plot(r, fun="log", main="log-survival curve")

plot(r, fun="event", main="cumulative events: f(y)=1-y")

plot(r, fun="cumhaz", main="cumulative hazard: f(y)=-log(y)=Lambda")

try( plot(r, fun="cloglog", main="complementary log-log plot") ) # f(y)=log(-log(y)), log-scale on the x-axis
On peut aussi avoir un (ou plusieurs) facteurs -- nous en reparlerons plus loin.
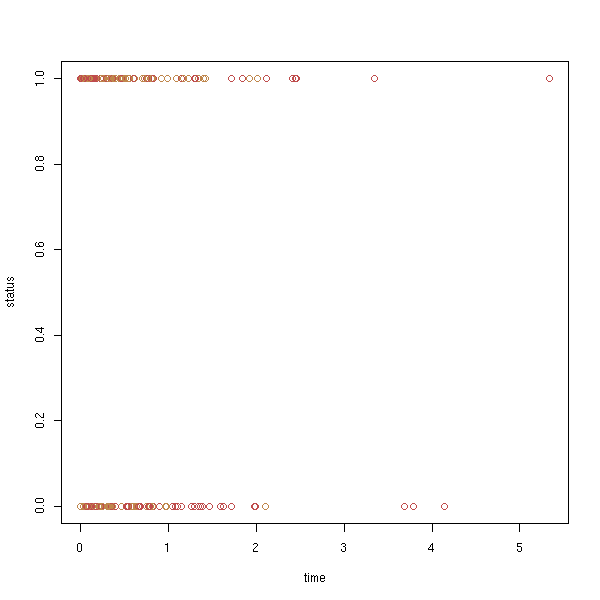
n <- 200 x1 <- rweibull(n,.5) x2 <- rweibull(n,1.2) f <- factor( sample(1:2, n, replace=T), levels=1:2 ) x <- ifelse(f==1,x1,x2) y <- rexp(n,1/mean(x)) s <- Surv(ifelse(x<y,x,y), x<=y) plot(s, col=as.numeric(f))
plot( density(s[,1][ s[,2] == 1 & f == 1]), lwd=3,
main="Analyse de la survie, un facteur" )
lines( density(s[,1][ s[,2] == 0 & f == 1]), lty=2, lwd=3 )
lines( density(s[,1][ s[,2] == 1 & f == 2]), col='red', lwd=3 )
lines( density(s[,1][ s[,2] == 0 & f == 2]), lty=2, col='red', lwd=3 )
legend( par("usr")[2], par("usr")[4], yjust=1, xjust=1,
c("non censored, f = 1", "censored, f = 1",
"non censored, f = 2", "censored, f = 2"),
lty=c(1,2,1,2),
lwd=1,
col=c(par('fg'), par('fg'), 'red', 'red') )

plot(survfit(s ~ f), col=as.numeric(levels(f)))
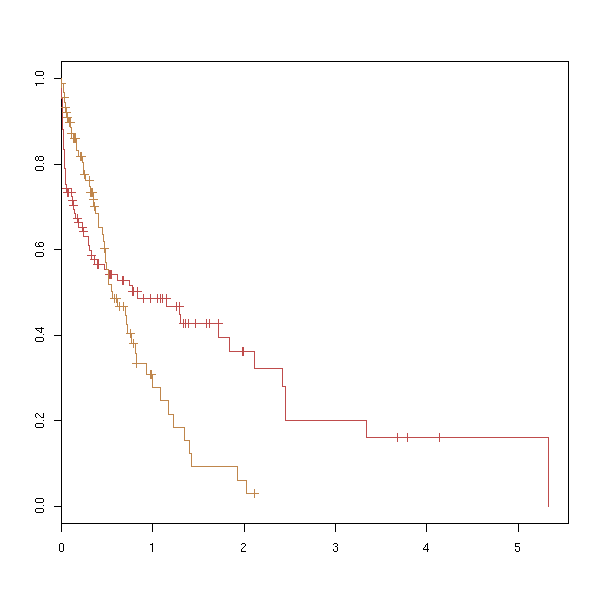
A FAIRE : avec deux facteurs survfit(s~f1+f2)
A FAIRE : et si f n'est pas un facteur ??? (mettre ça un peu plus loin, quand on parlera de régression d'une variable de survie).
A FAIRE : choisir deux exemples, l'un avec l'autre sans covariables, que l'on utilisera jusqu'à la fin de ce chapitre.
data(lung) x <- Surv(lung$time, lung$status) plot(x)

plot(survfit(x))


/image%2F4667470%2F20201115%2Fob_e77730_2ec8f0db-bdac-4bd6-a269-846fc8ce27b2.gif)
/image%2F4667470%2F20211129%2Fob_7729b4_983015ac-9f27-40d0-81d8-a8e5c0d21891.jpeg)